
Revista de Ciencias Ambientales (Trop J Environ Sci). (Enero-Junio, 2019). EISSN: 2215-3896. Vol 53(1)
DOI: http://dx.doi.org/10.15359/rca.53-1.7
Open Acces: www.revistas.una.ac.cr/ambientales
EMAIL: revista.ambientales@una.cr
Umaña-Castro J., Orozco-Cayasso S., Umaña-Castro R., Molina-Bravo R.
Identificación molecular y características fisiológicas de aislamientos de Trichoderma para el biocontrol de dos patógenos en la piña
Molecular Identification and Physiological Characteristics of Trichoderma Isolates for the Biocontrol of Two Pathogens in Pineapple
Josué Umaña-Castro1, Steffany Orozco-Cayasso2, Rodolfo Umaña-Castro3, Ramón Molina-Bravo4
[Recibido: 19 de julio 2018, Aceptado: 16 de octubre 2018, Corregido: 16 de noviembre 2018, Publicado: 1 de enero 2019]
Nota: Resultados parciales de este artículo fueron presentados en el I Congreso Centroamericano de Ciencias de la Tierra y el Mar, organizado por la Facultad de Ciencias de la Tierra y el Mar de la Universidad Nacional. 13-15 noviembre 2017, San José, Costa Rica.
Resumen
La contaminación ambiental por uso excesivo de agroquímicos ha dado lugar a la búsqueda de alternativas en la agricultura. Por esta razón, ha surgido el uso de agentes biológicos como alternativa para el combate de patógenos. Trichoderma es un agente biológico ampliamente utilizado, pero es difícil identificarlo a nivel de especie por sus características morfológicas. Además, Trichoderma forma complejos de especies, por lo que aumenta la importancia de ser correctamente identificado. Los productos biológicos también requieren ser fáciles de reproducir in vitro, generar conidios con alto porcentaje de germinación y demostrar actividad antagónica parciales agente causal. En este estudio, el objetivo fue determinar si las relaciones genéticas de 15 aislamientos de Trichoderma se asocian a 3 características deseables en productos de control biológico. Se analizaron 15 aislamientos monospóricos de Trichoderma nativos de la Región Huetar Norte de Costa Rica con efecto antagónico contra patógenos de la piña. Se midió la velocidad de crecimiento y el porcentaje de germinación. La velocidad de crecimiento promedio de los aislamientos a 25 ºC varió desde 0.90 a 1.20 mm · h-1 y el porcentaje de germinación entre 3 y 94 %. Para identificar las especies de este estudio, se secuenció el gen del factor de elongación de la traducción, TEF1-α y se identificaron 4 especies: T. reesei, T. spirale, T. asperellum/asperelloides y T. koningiopsis. Las distancias genéticas entre las especies se midieron utilizando 6 intermicrosatélites (ISSR). Las distancias genéticas mostraron una alta diversidad inter e intraespecífica. En condiciones controladas de laboratorio, el aislamiento 8a (T. reesei) mostró mayor efecto antagónico contra dos patógenos de la piña, donde Fusarium oxysporum tuvo un crecimiento de 30 % y Pectobacterium carotovorum un 63 % en presencia de Trichoderma. Un test de Mantel determinó que no hay correlación entre las distancias genéticas y las distancias euclidianas de los parámetros de crecimiento ni con la respuesta de antagonismo. Se concluyó que estas características no son especie dependientes; sin embargo, los aislamientos mostraron alta diversidad dentro de un área geográfica poco amplia con características de interés biocontrolador.
Palabras clave: Combate biológico; Fusarium oxysporum; intermicrosatélites, Pectobacterium carotovorum; TEF1-α.
Abstract
Environmental pollution associated with the excessive use of pesticides has prompted the need for alternative practices in agriculture, such as the application of biological agents for controlling plant pathogens. Trichoderma is an agent that is amply used for biocontrol, however the fungus is difficult to identify at the species level strictly by morphological examination. Additionally, the genus forms species complexes, which can make species identification much more challenging. Effective biocontrol products have to be easily reproducible in vitro, generate conidia with high germination rates, and demonstrate effective antagonistic activity against a target pathogen. The objective of this study was to determine if these desirable biocontrol traits are correlated with the genetic relationships of 15 Trichoderma isolates. We analyzed 15 monosporic Trichoderma isolates native to the northern region of Costa Rica with known antagonistic activity against pathogens associated to pineapple. We measured growth rate and germination percentage. Mean growth rates varied from 0.90 to 1.20 mm · h-1 at 25 ºC. Germination percentage varied greatly from 3 % to 94 %. Species were identified by sequencing the translation elongation factor, TEF1-α, gene and four species were identified: T. reesei, T. spirale, T. asperellum/asperelloides and T. koningiopsis. Genetic distances within and among species were measured using six intermicrosatellites (ISSR). These distances showed high inter and intraspecific diversity. Under a control lab environment, isolate 8a (T. reesei) showed the largest antagonistic effect against two pineapple pathogens, where there was a 30 % growth inhibition of Fusarium oxysporum and a 63 % growth inhibition of Pectobacterium carotovorum. A Mantel test showed there was no correlation between genetic distances and Euclidean values for growth parameters and antagonistic response. We concluded that these traits were not species-dependent, however, these isolates had high genetic diversity in a small, narrow geographical area with important biocontrol traits of interest.
Keywords: Biological control; Fusarium oxysporum; intermicrosatélites; Pectobacterium carotovorum; TEF1-α.
1.Introducción
Existen diferentes enfoques que pueden ser usados para prevenir, mitigar o manejar enfermedades de las plantas. Más allá de las buenas prácticas agronómicas y hortícolas, los grupos productores a menudo se basan en gran medida en los fertilizantes y productos agroquímicos. Sin embargo, la contaminación ambiental causada por el uso excesivo de estos ha dado lugar a considerables cambios en las actitudes de las personas hacia el uso de plaguicidas en la agricultura (Pal & McSpadden, 2006). Por lo anterior, el uso de agentes de combate biológico se ha convertido en una opción para el manejo de enfermedades de los cultivos y surge el uso de biocontroladores de patógenos como Trichoderma.
Desde los primeros estudios sobre organismos antagonistas hasta el presente, el hongo del género Trichoderma ha sido uno de los organismos más investigados y se ha usado para el combate biológico de diferentes patógenos como Fusarium oxysporum, Botrytis cinerea, Rhizoctonia solani, Sclerotium rolfsii, Pythium sp., Alternaria sp. (Infante, Martínez, González, & Reyes, 2009). Vásquez (2009) obtuvo resultados favorables con un aislamiento de Trichoderma proveniente de una finca piñera en la zona de Venecia de San Carlos en Costa Rica, donde logró una colonización del 85 % sobre F. oxysporum mediante pruebas de antagonismo con la técnica de cultivos duales y potencial fungistático. Además de ser utilizado para el combate biológico de hongos fitopatógenos, Trichoderma funciona para el manejo de Pectobacterium carotovorum (anteriormente Erwinia sp.) en el cultivo de la piña (Vargas, 2011), con lo cual demuestra que el hongo del género Trichoderma funciona también como antagonista de bacterias.
Chaverri, Branco, Jaklitsch, Degenkolb y Samuels (2015) evaluaron 4 productos comerciales de Trichoderma reportados como T. harzianum para combate biológico. Después de aislar las cepas de estos productos, encontraron e identificaron 14 especies diferentes de Trichoderma; sin embargo, ninguna fue identificada como T. harzianum. Esto indica que existen mezclas de especies de Trichoderma en productos comerciales. Martínez, Infante y Peteira (2015) mencionan que la taxonomía basada en caracteres morfológicos no es suficiente para diferenciar las especies del género Trichoderma, debido a la gran similitud que existe entre ellas. Por lo tanto, la aplicación de técnicas moleculares es necesaria para la identificación y clasificación de este género. Además de la identificación de especie, es importante determinar las relaciones genéticas entre aislamientos de una misma especie para ver diversidad no solo entre especies, sino también dentro de las especies. Esto se puede realizar por medio de marcadores moleculares, tales como los intermicrosatélites, que han demostrado ser útiles para medir relaciones genéticas entre cepas de Trichoderma (Consolo, Mónaco, Cordo, & Salerno, 2012).
Es de importancia tener información sobre las condiciones óptimas para la germinación y crecimiento de cada hongo, porque brindaría una idea de su crecimiento in vitro para la elaboración de un producto de combate biológico a nivel masivo. Guigón-López et al., (2010), en análisis moleculares realizados a cepas de Trichoderma aisladas a partir de suelo, demostraron que la tasa de crecimiento del micelio es importante para pronosticar la capacidad de biocontrol de las cepas.
En un estudio previo elaborado por personal del Laboratorio de Fitopatología de la Universidad Nacional se recolectaron 15 aislamientos de Trichoderma provenientes de fincas piñeras en la zona Norte de Costa Rica, donde se demostró, de forma in vitro, su efecto antagónico contra F. oxysporum y P. carotovorum. Estos se preservaron en la colección de hongos de este laboratorio; sin embargo, es de importancia caracterizarlos genética y morfológicamente.
En este estudio esos 15 aislamientos de Trichoderma se caracterizaron y se determinaron sus relaciones genéticas por medio de 2 sistemas de marcadores moleculares y 2 características fisiológicas. Se analizó el crecimiento diametral y el porcentaje de germinación (viabilidad) de esporas de cada uno de ellos. Pruebas previas a este estudio demostraron que estos aislamientos tienen actividad antagónica in vitro contra los patógenos de la piña, F. oxysporum y P. carotovorum, por lo tanto, se realizaron distintos análisis para elucidar las especies asociadas a secuencias de marcadores genéticos, sus relaciones genéticas y fisiológicas.
2.Metodología
2.1Origen y reproducción de los aislamientos
Se analizaron 13 aislamientos monospóricos de Trichoderma de la colección de hongos del Laboratorio de Fitopatología de la Universidad Nacional (UNA) de Costa Rica, provenientes de la zona norte de Costa Rica, aislados a partir de suelo en fincas piñeras y parches de bosque aledaños a las plantaciones en Pital de San Carlos, río Cuarto de Grecia, Sarapiquí y Muelle de San Carlos (Apéndice 1). Los aislamientos se reactivaron en placas petri con PDA (papa dextrosa agar) acidificado con ácido láctico al 25 % y se incubaron a temperatura ambiente hasta el desarrollo de las colonias fungosas y su esporulación. Una vez esporuladas, se realizaron 3 réplicas en las que se tomó el micelio de cada cepa para ser colocado en nuevas placas Petri; nuevamente se esperó su esporulación libre de otros microorganismos (French & Herbert, 1980).
2.2Velocidad del crecimiento micelial y porcentaje de germinación (viabilidad)
Se midió el crecimiento diametral durante 72 h; para esto, se siguió un protocolo modificado basado en el descrito por Díaz y Lecuona (1995). Se sembró un cuadro de 1 cm de PDA con el aislamiento purificado en el extremo del plato petri con medio PDA y se incubó a 25 ºC. Se tomaron 3 mediciones por aislamiento, a las 24, 48 y 72 h. Se realizaron 3 repeticiones por cada aislamiento.
Se determinó el porcentaje de germinación (viabilidad) de esporas en agar agua aplicando un protocolo modificado basado en aquel descrito por Tapsoba y Wilson (1997). Se colocaron 5 alícuotas de 5µl de una suspensión de conidios con una concentración de 1 x 106 conidios · mL-1 en medio agar agua para cada aislamiento. Se incubaron a temperatura ambiente y a las 16 horas se agregó azul de lactofenol para detener la germinación. Posteriormente, se cortó la porción de agar que contenían las alícuotas de la suspensión de conidios y se colocó sobre un portaobjetos cubierto con un cubreobjetos. Se registró el número de conidios germinados. Se consideraron germinados aquellos conidios cuyo tubo germinativo fue 2 veces mayor a su diámetro. Se realizaron 5 repeticiones por cada aislamiento. A las 16 horas se calculó el porcentaje de germinación y se contó tanto el número total de conidios como los conidios germinados en el campo visual del microscopio a 40X.
2.3Identificación de especies y análisis molecular
El ADN de los aislamientos se extrajo a partir de micelio esporulado siguiendo el protocolo de Hoyos-Carvajal, Chapparro, Ambramsky, Chet y Orduz (2008) y se visualizó por medio de electroforesis en geles de agarosa al 1 %, para observar calidad y cantidad. Se utilizó buffer de carga con Gel Red™ (Biotium) al 0.3 % para la tinción. La cantidad del ADN de cada muestra se comparó con ADN lambda como estándar. La intensidad del fragmento se midió por medio del programa ImageJ, el cual cuantificó los pixeles digitales contra el estándar.
Se amplificaron 2 regiones del factor de elongación de la traducción (TEF1-α) para la identificación de los aislamientos. Se utilizaron 2 pares de cebadores reportados por Semuels, Ismaiel, de Sauza, y Chaverri (2012) y Chaverri et al., (2015): EF-728M (5’- CAT YGA GAA GTT CGA GAA GG) y Ef2 (5’- GGA RGT ACC AGT SAT CAT GTT); y EF700F (5’- TCT ACC AGT GCG GTG GTA) y TEF1R (5’- GCC ATC CTT CGA ACC AGC). Las PCR se realizaron en las siguientes condiciones de termociclado: desnaturalización inicial a 94 °C por 5 min; 36 ciclos de 94 °C con duración de 1 min, luego a 56 °C por 1 min y 72 °C por 1 min; finalizado por una extensión a 72 °C por 5 min. La calidad de los productos de PCR fueron verificados en un gel de agarosa al 1% y se repitió todo el proceso de la PCR hasta obtener un producto en abundancia del tamaño esperado de 600 pb. Se enviaron 30 productos de PCR a la empresa Macrogen® Corea para su purificación y secuenciación por método Sanger en ambas direcciones de la cadena.
Se obtuvieron secuencias consenso a partir de las secuencias bidireccionales de las 2 regiones del TEF-1α con el programa Geneious® versión R٨ (Biomatters Ltda). Para determinar la identidad primaria de cada aislamiento, ambas secuencias del TEF-1α se compararon con las accesiones depositadas en la base de datos del National Center for Biotechnology Information (NCBI) mediante el programa de búsqueda en línea BLAST (Basic local Alignment Search Tool). Se realizaron alineamientos múltiples de similitud genética con todas las secuencias locales y accesiones de las bases de datos utilizando el programa en línea MAFFT 7.0 (http://mafft.cbrc.jp/alignment/server), con el método FFT-NS-i y el parámetro 1PAM / k = 2 determinado para secuencias relacionadas. Se utilizó el programa Gblocks (Castresana, 2000) para seleccionar bloques sin alineamientos erróneos, ni regiones divergentes. Se construyó un árbol de posicionamiento taxonómico basado en el TEF-1α, con la utilización del programa de acceso libre raxmlGUI v.7.4.2 (Stamatakis, Ludwig, & Meier, 2005), se empleó el modelo de sustitución de nucleótidos GTR-GAMMA. Se calculó el porcentaje de confianza por medio del método Bootstrap con 2000 iteraciones. El análisis incluyó secuencias de Trichoderma de clima tropical y templado depositadas en el NCBI, así como 2 secuencias de F. oxysporum como grupos externos.
2.4Relaciones genéticas
Se realizó una prueba preliminar con 20 cebadores ISSR reportados por Consolo et al. (2012) y Verma et al. (2009). Se realizaron reacciones con los 20 cebadores hasta obtener 6 que generaron productos polimórficos. Se eligieron los 6 cebadores que aportaron más información con base en la claridad y cantidad de fragmentos generados. Las PCR se realizaron con las siguientes condiciones en un volumen final de 25 µL: un paso de desnaturalización a 94 °C durante 7 min; seguido por 33 ciclos a 94 °C durante 60 s, 48 °C durante 75 s y 72 °C durante 4 min; y una etapa de extensión final de 72 °C durante 7 min. Los productos de PCR se separaron en geles de agarosa al 1 % teñidos con Gel Red® con un tiempo de movilización de 3 a 4 h a 100 V y fueron digitalizados en un fotodocumentador BioDoc-It (UVP).
Para las relaciones genéticas con los ISSRs se generó una matriz binaria de los alelos. Se estructuró una matriz de distancias genéticas y se determinó la diversidad alélica por aislamiento, así como un análisis de variación molecular (AMOVA) con el programa de libre acceso GenAlex (Peakall & Smouse, 2006). Se generó y se calcularon las distancias del dendrograma por medio del método Neighbor Joining para representar diferencias de cepas similares con el programa Splits Tree (Huson,1998). Los porcentajes de confiabilidad se calcularon por medio de un Bootstrap con 2 000 iteraciones.
2.5Análisis estadísticos
Se realizó una regresión lineal para cada cepa, donde la pendiente de la función se consideró la velocidad del crecimiento (mm · h-1). Los datos no cumplieron con las suposiciones de normalidad y varianzas homogéneas del análisis de varianza (ANOVA) según un test de Bartlett y Shapiro-Wilk. Por lo tanto, se aplicó un test Kruskall-Wallis para datos no paramétricos con comparaciones múltiples y un ajuste del valor p con el método de Holm. Diferencias significativas se declararon cuando p < 0.05. Los datos de porcentaje de germinación se transformaron por medio de la función arco seno. Se aplicó un ANOVA de un factor con un análisis post hoc de Tukey, donde diferencias significativas se declararon cuando p < 0.05. Los análisis se realizaron por medio del programa R (R Core Team, 2016).
Se probó la correlación entre la distancia genética y las mediciones fisiológicas de crecimiento diametral (velocidad de crecimiento en mm · h-1) y porcentaje de germinación, así como la actividad antagonista (dato proporcionado por el Laboratorio de Fitopatología) por medio de un test de Mantel en el programa GenAlEx. Se realizó un análisis de correlaciones entre las distancias genéticas y los datos fenotípicos utilizando el paquete vegan para el programa estadístico R (Oksanen et al., 2017). Un análisis de componentes principales de los datos morfológicos se ejecutó por medio del comando prcomp y fue graficado con el comando autoplot del paquete ggplot2 en R (Wickham, 2016).
3.Resultados
3.1Características morfológicas
Todos los aislamientos de Trichoderma cubrieron casi la totalidad del plato petri en 72 horas. Los aislamientos 11fa, 8a, 6a y 11c tuvieron comportamientos similares, pero se determinaron diferencias estadísticamente significativas (p < 0.05) con respecto a los 9a, Ia, H1, 13c y 1c (Cuadro 1). Por lo anterior, los aislamientos 11fa, 8a, 6a y 11c fueron los que alcanzaron una mayor velocidad de crecimiento con un promedio > 1 mm•h-1. Las velocidades de crecimiento variaron notablemente (entre 0.74 y 1.20 mm•h-1), sin embargo, los aislamientos 11fa, 8a, 6a y 11c mostraron la mayor capacidad de crecimiento micelial (Figura 1). El porcentaje de germinación o viabilidad de cada aislamiento (Cuadro 1) varió entre 3 % y 90 %, a pesar de estar en condiciones similares. Los aislamientos 11fb, 13a, 13c y 6a tuvieron una germinación de más del 80 %. El porcentaje de germinación aportó la mayoría de la variabilidad para distinguir aislamientos (Figura 1).
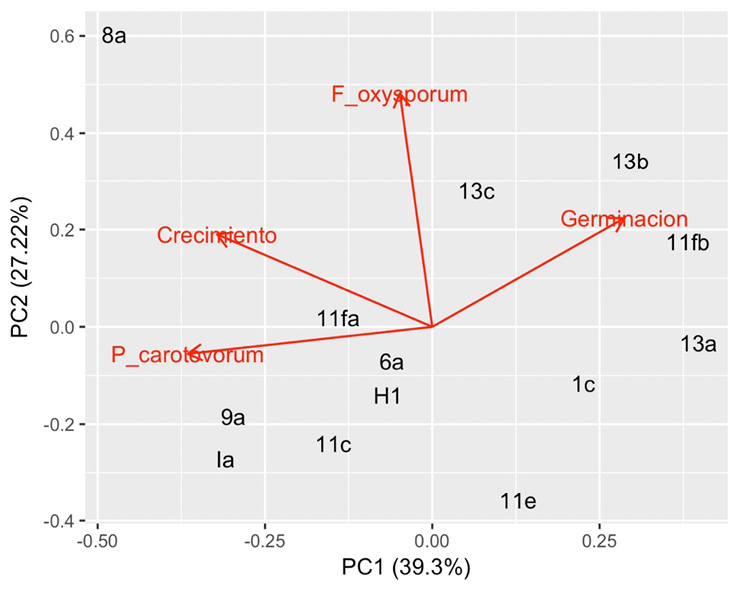
Figura 1. Análisis de componentes principales con valores ajustados para 4 características fisiológicas en 15 aislamientos de Trichoderma: velocidad de crecimiento (Crecimiento), porcentaje de germinación de esporas (Germinacion) y la inhibición del crecimiento por la actividad antagonista contra Fusarium oxysporum (F_oxysporum) y Pectobacterium caratovorum (P_caratovorum). Flechas rojas indican el peso separativo de cada característica.
3.2Antagonismo contra F. oxysporum y P. caratovorum
Los experimentos in vitro mostraron que Trichoderma spp. tuvo un efecto antagónico sobre F. oxysporum. En comparación con un testigo, los aislamientos que ejercieron una mayor actividad antagónica fueron la 8a, 13b y 13c. Todos mostraron ser promisorios impidiendo el desarrollo del patógeno, sin embargo, F. oxysporum en presencia del aislamiento 8a presentó 17.30 mm de crecimiento al término de 5 días en comparación con el testigo que creció 57.84 mm (Este testigo contenía solo a F. oxysporum). Estas diferencias fueron estadísticamente significativas (p < 0.05; Cuadro 1).
Cuadro 1. Tests de Kruskall-Wallis de características de crecimiento micelial (mm · h-1) y efecto antagónico de los aislamientos de Trichoderma sobre Fusarium oxysporum y y ectobacterium carotovorum. Análisis de Tukey realizado a los porcentajes de germinación tomado a las 16 h de desarrollo. Valores para los límites inferiores recalcados en gris. Valores para los límites superiores recalcados en negrita.
|
Código |
Crecimiento |
Germinación (%) |
Antagonismo (mm) |
|
|
F. oxysporum |
P. carotovorum |
|||
|
Testigo |
*57.84 |
**87.63 |
||
|
11fa |
1.20 |
61.19 |
32.81 |
65.59 |
|
8a |
1.17 |
34.68 |
17.30 |
55.38 |
|
6a |
1.02 |
88.34 |
33.14 |
57.02 |
|
11c |
1.02 |
3.68 |
32.02 |
66.02 |
|
9a |
0.99 |
15.61 |
30.34 |
56.10 |
|
Ia |
0.93 |
14.29 |
30.98 |
62.92 |
|
H1 |
0.97 |
16.77 |
30.00 |
65.96 |
|
13c |
0.90 |
83.73 |
23.80 |
59.93 |
|
1c |
0.90 |
78.23 |
32.49 |
66.70 |
|
11fb |
0.87 |
94.01 |
27.24 |
71.27 |
|
13b |
0.90 |
56.78 |
22.01 |
75.22 |
|
13a |
0.80 |
91.07 |
30.26 |
68.72 |
|
11e |
0.74 |
15.41 |
31.08 |
64.70 |
* Aislamiento puro de F. oxysporum.
** Aislamiento puro de P. carotovorum.
Los experimentos in vitro mostraron que Trichoderma spp. también tuvieron un efecto antagónico sobre P. carotovorum. En comparación con un testigo, los aislamientos que ejercieron una mayor actividad antagónica fueron la 8a, 13b y 13c (Cuadro 1). Todas mostraron ser promisorias al impedir el desarrollo por completo del patógeno, sin embargo, P. carotovorum en presencia del aislamiento 8a presentó diferencias significativamente mayores (p < 0.05) que los demás, con un crecimiento de 55.38 mm del patógeno en comparación con un testigo de 87.63 mm a los 5 días. Estos análisis de Trichoderma sobre F. oxysporum y P. carotovorum mostraron que el aislamiento 8a desempeñó un mayor efecto antagónico contra ambos patógenos.
3.3Identificación de especies y análisis molecular
El Cuadro 2 muestra que del total de aislamientos secuenciados se obtuvieron 4 especies de Trichoderma, las cuales fueron: T. spirale, T. asperellum, T. koningiopsis y T. reesei. Todas las especies obtuvieron más de un 95 % de identidad en comparación con secuencias depositadas en la base de datos de NCBI.
Cuadro 2. Resultados de la secuenciación de los fragmentos del gen TEF-1α (500-600 pb) por medio de la combinación de los cebadores EF-728M y Ef2, y EF700F y TEF1R (Chaverri, Branco, Jaklitsch, Degenkolb y Samuels, 2015; Samuels, Ismaiel, de Sauza y Chaverri, 2012) para aislamientos de Trichoderma. Porcentajes de identidad (%) fueron calculados por medio de la herramienta en línea BLAST Nucleotide de National Center for Biotechnology Information (NCBI). Similitud en pares de bases (pb) de las secuencias originales con las depositadas en la NCBI.
|
Código |
Similitud (%) |
Especie reportada por NCBI |
Identidad (%) |
|
13a |
531pb/525pb |
T. spirale |
99 |
|
11e |
541pb/526pb |
T. spirale |
97 |
|
13c |
531pb/523pb |
T. spirale |
98 |
|
11fb |
581pb/553pb |
T. asperellum |
95 |
|
13b |
579pb/576pb |
T. asperellum |
99 |
|
H1 |
584pb/577pb |
T. asperellum |
99 |
|
9a |
570pb/560pb |
T. koningiopsis |
98 |
|
1c |
552pb/544pb |
T. koningiopsis |
99 |
|
6a |
486pb/467pb |
T. koningiopsis |
96 |
|
Ia |
570pb/558pb |
T. konongiopsis |
98 |
|
11fa |
574pb/556pb |
T. koningiopsis |
97 |
|
11c |
555pb/548pb |
T. koningiopsis |
99 |
|
8a |
530pb/526pb |
T. reesei |
99 |
En la Figura 2 se presenta un árbol filogenético con las especies obtenidas mediante secuenciación del (TEF-1α). Se corroboró el posicionamiento taxonómico de las cepas analizadas, las cuales se agruparon con cepas de la misma especie. En el clado 1 se encontró el aislamiento 8a (T.reesei), la cual se agrupó con accesiones de la misma especie de diferentes lugares del mundo, incluida Hipocrea jecorina, la fase asexual de T. reesei. El clado 2 corresponde a T. spirale, el clado 3 a T. asperellum y T. asperelloides y el clado 4 a T. koningiopsis. Todos los anteriores también se agruparon con accesiones de su misma especie en donde el valor bootstrap fue entre el 70 y el 100 %.
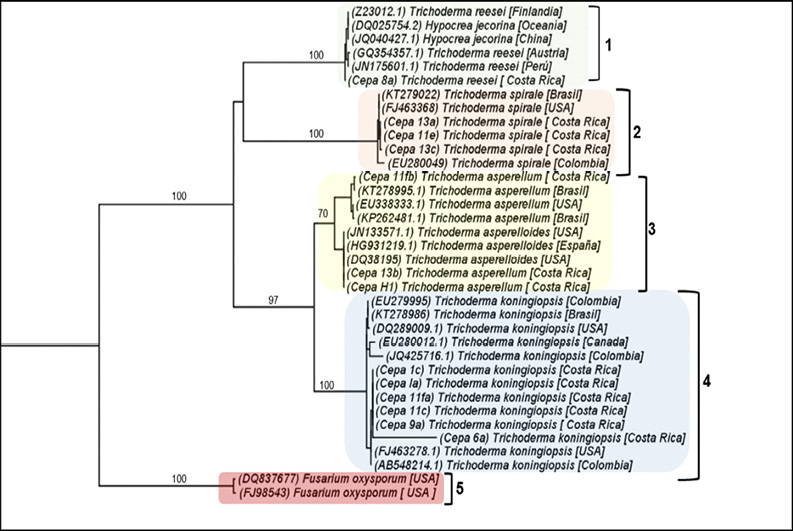
Figura 2. Topología filogenética de máxima verosimilitud basada en las secuencias parciales del gen del factor de elongación de la traducción 1-α (TEF-1α) que incluye aislamientos de Trichoderma aisladas de la zona norte de Costa Rica (las utilizadas en este estudio) y secuencias obtenidas a partir de la base de datos del NCBI.
3.4Relaciones genéticas
Según el dendrograma de distancias genéticas (Figura 3) calculado por los marcadores ISSRs, el clado 1 (T. spirale) colocó los aislamientos 13a y 13c estrechamente relacionados, mientras que 11e, a pesar de que estos 3 fueron identificados como T. spirale, reveló una separación más notable. En la Figura 3, el clado 2 (T. asperellum) mostró las cepas 13b y H1 como estrechamente relacionadas, mientras que la 11fb se separó de las anteriores y fueron la misma especie. El clado 3 (T. koningiopsis) presentó una mayor similitud entre los aislamientos que los demás clados, sin embargo, los aislamientos 11fa y Ia presentaron diferencias en comparación con 1c y 11c. Estas también se aislaron de la zona norte de Costa Rica.
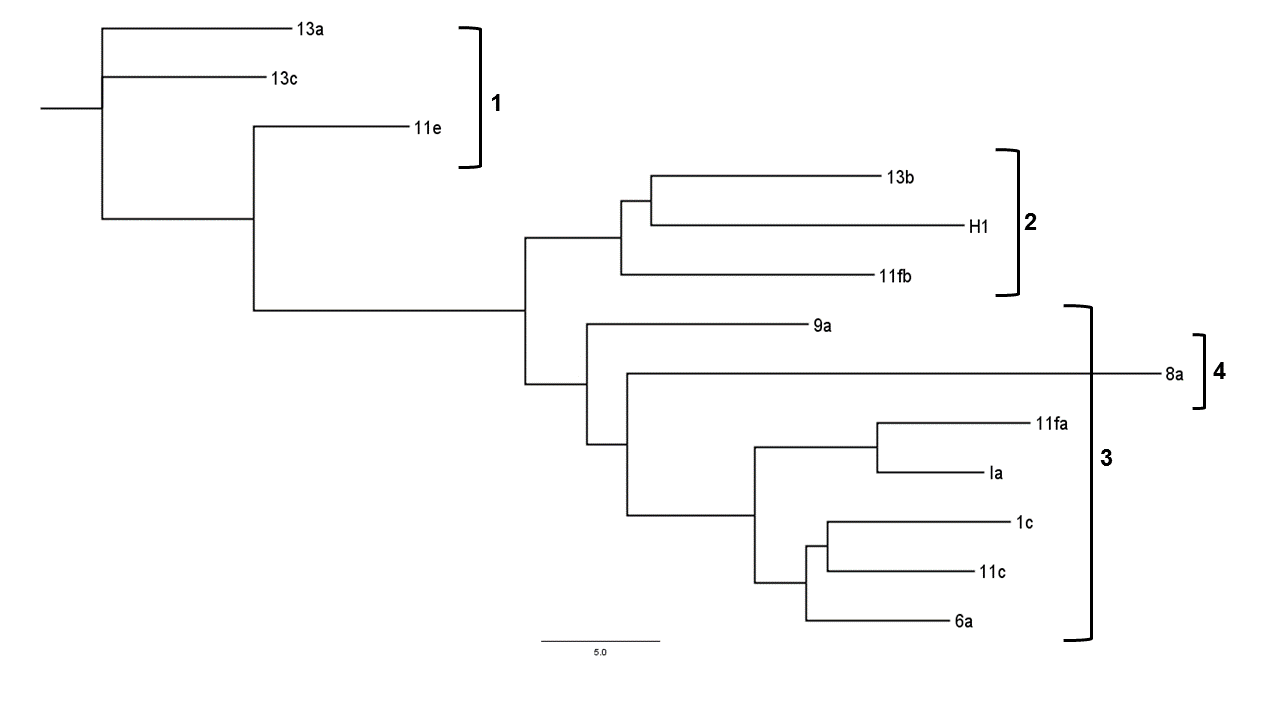
Figura 3. Dendrograma de distancias (relaciones) genéticas analizadas por medio de 6 marcadores moleculares tipo ISSR y el método Neighbor Joining (NJ) de aislamientos de Trichoderma de la zona norte de Costa Rica. Especies identificadas por medio de comparaciones de la base de datos NCBI de las regiones variables del gen del factor de elongación de la traducción (TEF1-α). Especies indicadas en llaves numeradas: T. spirale (1), T. asperellum (2), T. koningiopsis (3) y T. reesei (4).
Por último, la cepa identificada como T. reesei presentó similitudes con el clado 3; sin embargo, fue el aislamiento genéticamente más distante. Se realizó un test de Mantel para correlacionar las distancias genéticas con las coordenadas geográficas de recolección de cada aislamiento, el cual no fue significativo, por lo tanto, la distancia geográfica entre especies no determina su variabilidad.
Cuando se calcularon las distancias euclidianas de los datos morfológicos (velocidad de crecimiento), las pruebas de Mantel no encontraron ninguna correlación con las distancias genéticas (p < 0.86). Tampoco hubo correlación entre las características morfológicas (p < 0.109). Sin embargo, la velocidad de crecimiento presentó una leve correlación positiva con la diversidad genética. Aun así, la correlación no fue significativa (p < 0.26). Esta correlación podría demostrarse con confiabilidad utilizando un mayor número de muestras para el análisis. Al calcular las distancias euclidianas de los datos de efecto antagónico de Trichoderma spp. sobre F. oxysporum y P. carotovorum, las pruebas de Mantel tampoco encontraron correlación con las distancias genéticas (p < 0.34) ni entre el desempeño de las 2 actividades antagónicas de ambos patógenos.
3.5Análisis estadísticos
Al comparar el porcentaje de germinación y la velocidad de crecimiento, no hubo ningún tipo de correlación (p < 0.86) con las distancias genéticas. De la misma manera, un porcentaje de más del 90 % de germinación no coincidió con una tasa de crecimiento alta y viceversa. Tampoco hubo correlación entre las distancias genética y el antagonismo (p < 0.34). Chavarría (2016) en su estudio obtuvo resultados similares, ya que el porcentaje de viabilidad en aislamientos de Trichoderma fue de un 100 % a las 72 horas, mientras que en algunas su velocidad de crecimiento fue de 0.70 mm·h-1 y en otras fue mucho menor correspondiente a 0.40 mm·h-1. No hubo correlación significativa entre las distancias de disimilitud genética y las distancias geográficas, lo cual significa que la distancia geográfica no fue un factor de determinación de la diferenciación genética, aunque hubo alta variabilidad entre especies (p < 0.001).
4.Discusión
4.1Velocidad del crecimiento micelial y porcentaje de germinación (viabilidad)
El aislamiento 8a (T. reesei), a pesar de mostrar la mayor capacidad antagónica, no tuvo la mayor velocidad de crecimiento de micelio (1.17 mm · h-1) y porcentaje de germinación (34.68 %). Steyaert et al. (2013) mencionaron que la producción de conidios de Trichoderma se estimula por cambios ambientales muy complejos que interactúan entre sí para lograr una reproducción exitosa. En este estudio los porcentajes más altos de germinación de conidios corresponden a las cepas 13a y 13c ubicadas en el clado 1, el cual estuvo genéticamente distante al aislamiento 8a. También Valencia-Abellos y Castro-Caicedo (2004) midieron el crecimiento micelial de Trichoderma, el cual fue más rápido en temperaturas entre 26–30 ºC; sin embargo, el porcentaje de germinación (82 %) fue mayor cuando se incubó a 21 ºC a una concentración de 1x103 conidios · mL-1 observado a las 17 horas. Lo anterior sugiere que cada mecanismo de reproducción es diferente y que a cada aislamiento se le deben dar las condiciones adecuadas para su desarrollo de acuerdo con la especie, pero tomando en cuenta que estos parámetros varían a nivel del individuo. En este estudio, a todos los mecanismos se les dieron las mismas condiciones de temperatura ambiente (25 ºC) por lo que no se puede atribuir a la especie características de crecimiento, germinación y antagonismo debido a que no se encontró correlación. Adicionalmente, se reporta que especies como T. asperellum y T. harzianum crecen mejor a temperaturas entre 27–30 ºC (Watanabe et al., 2005), y un máximo de 35 °C (Guigón-López et al., 2010).
4.2Análisis genéticos
Se obtuvieron 4 especies: T. reesei/H. jecorina, T. spirale, T. asperelum/asperelloides y T. koningiopsis. Según Atanasova et al. (2010), T. reesei/H. jecorina ha sido encontrado en suelos tropicales del sureste de Asia, Suramérica y regiones del sur del Pacífico, donde su reproducción es ideal entre los 25–30 ºC. Druzhinina, Komon, Atanasova, Seidl y Kubicek (2010) demostraron que T. reesei es un antagonista moderado contra los patógenos Sclerotinia sclerotiorum, Alternaria alternata y Botritis cinerea y que se ha encontrado también en Centroamérica y Suramérica. Trichoderma spirale es un hongo cosmopolita, el cual ha sido encontrado en el tronco de plantaciones de cacao en San Vito de Coto Brus, Puntarenas, Costa Rica y su crecimiento se desarrolla mejor en temperaturas entre 25–30 ºC (Chaverri, Castlebury, Samuels, & Geiser, 2003). Trichoderma asperelum/asperelloides ha sido encontrada por Guigón-López et al., (2010) en suelos de la rizosfera en un cultivo de chile en Chihuahua, México, en donde su temperatura óptima de crecimiento es de 27–30 ºC. Trichoderma koningiopsis, según Jaimes, Moreno y Cotes (2009), ha demostrado ser eficaz en el control de F. oxysporum y también ha demostrado ser un inductor de defensa. También, tuvo un crecimiento mayor al 50 % a 21 ºC en comparación a aislamientos de T. harzianum a esa misma temperatura (Sánchez, Barrera, Reybet, & Sosa, 2015).
Las especies de Trichoderma tienen una amplia plasticidad ecológica, lo que implica encontrar variabilidad genética en ellas, en donde factores bióticos y abióticos como temperatura, pH, humedad relativa, iluminación y sustratos juegan un papel muy importante en el comportamiento del hongo (Martínez et al., 2015). Esto nos indica la importancia de conocer el comportamiento y hábitat de cada uno para tomar decisiones en cuanto a la escogencia de cepas con actividad antagónica. En la Figura 3 se demostró la alta variabilidad presente en cepas de Trichoderma de la misma especie analizada en este estudio, aisladas de un clima tropical que prevalece en Costa Rica. A pesar de que los hongos fueron aislados a partir de ambientes similares y a distancias relativamente cortas entre ellas (a una circunferencia menor a 50 km), se observó una variabilidad entre especies.
El aislamiento 8a (T. reesei) presentó un mejor desempeño en cuanto a efecto antagónico en condiciones in vitro contra los 2 patógenos de la piña, donde F. oxysporum tuvo un crecimiento de 30% en presencia de Trichoderma y P. carotovorum. Además de ser uno de los mejores antagonistas, este aislamiento (8a) fue genéticamente distante a los demás en cuanto a variabilidad entre especies y aislamientos (Figura 3).
La correcta identificación molecular de aislamientos monospóricos es de suma importancia para así tener certidumbre de la composición de un producto. Además de su identificación, Martínez et al., (2015) mencionaron la importancia de explorar, caracterizar y explotar estas variaciones genéticas entre especies para así lograr realizar una selección adecuada de las cepas promisorias para el combate de enfermedades. En este estudio se logró evidenciar esas variaciones genéticas entre aislamientos. La variación genética de Trichoderma no dependió de las distancias geográficas de los aislamientos. Este fenómeno se ha observado también en Metarhizium, otro miembro del orden Hypocreales (Bidochka, Kamp, Lavender, Dekoning, & De Croos, 2001). Bidochka et al. (2001) sugirieron un cambio de paradigma sobre el mayor factor que determina la diversidad genética de Metarhizium, el cual se centra en factores ambientales similares sin importar la distancia geográfica. También se encontró que las características deseables analizadas para el combate biológico no dependieron de su especie o de sus relaciones genéticas. Esto, debido a las diferencias de comportamiento en cuanto a crecimiento y efecto antagónico dentro de las mismas especies. Sin embargo, todo producto que se aplique debe estar correctamente identificado para así tener un panorama de cual sería su posible comportamiento de acuerdo con los factores ambientales de la zona de aplicación.
Howell (2003) mencionó que los mecanismos de biocontrol de Trichoderma pueden funcionar de manera distinta en condiciones de laboratorio y en el campo debido a que factores como el tipo de suelo, temperatura, pH, humedad del ambiente, la planta, el suelo y otros aspectos de la microflora influyen en su comportamiento. También la interacción con la rizosfera y los microorganismos presentes en el ecosistema juegan un papel importante en el crecimiento y antagonismo de estos hongos, ya que los exudados de las plantas proporcionan alimento para la actividad de microorganismos del suelo, germinación y proliferación de patógenos que a la vez sirven de alimento para hongos antagonistas (Mukerji, Manoharachary & Singh, 2006), lo anterior evidencia la importancia de llevar estos análisis al campo para ser evaluados en una segunda etapa.
En este estudio no todas las características deseables para un producto biológico se encontraron en un mismo aislamiento, por ejemplo, se hallaron rangos de germinación entre 10 y 90 %, velocidades de crecimiento entre 0.74 y 1.20 mm·h-1 y porcentajes de antagonismo sobre los patógenos de 30 a 63 %. Existen alternativas para obtener todas estas características deseables en un solo aislamiento, por ejemplo, brindar las condiciones de reproducción más favorables (Howell, 2003). También esto se puede lograr mediante la ingeniería genética. Existen varios genes involucrados en estos procesos, por ejemplo, el gen tac1 se relaciona con el crecimiento, germinación, micoparasitismo y metabolismo secundario de Trichoderma en T. virens (Mukerji et al., 2006). Estos genes deben estudiarse más a profundidad en caso de recurrir a las técnicas de ADN recombinante como medio para obtener un aislamiento con capacidades deseables en cada una de sus etapas de reproducción.
5.Conclusiones
Se concluye que no hay correlación entre las características de crecimiento, efecto antagónico y distancias genéticas. A raíz de la identificación de diferentes especies de Trichoderma provenientes del cultivo de piña fue posible detectar los aislamientos con mayor efecto antagónico. Con dicha información, se pueden brindar las condiciones adecuadas para cada etapa del desarrollo, principalmente la esporulación. Se evidenció una variabilidad genética y morfológica significativa entre aislamientos recolectados a distancias geográficas relativamente cercanas. Es necesario realizar pruebas in vitro para determinar cuál es el mecanismo que utiliza el aislamiento T. reesei (8a) contra la bacteria y contra el hongo fitopatógeno, ya sea competición, antibiosis o micoparasitismo. Al identificar las especies de Trichoderma y posterior a determinar su efecto antagónico bajo condiciones controladas de laboratorio, según su comportamiento y hábitat de recolección, se deben probar en campo para observar su interacción con microorganismos de la raíz, suelo y planta, y así tomar la decisión final de cuáles, dentro de estas mismas especies, son los aislamientos adecuados para un combate biológico de distintos patógenos. Estos hallazgos beneficiarán a instituciones o empresas interesadas en elaborar biopesticidas para el combate biológico de enfermedades en cultivos.
6.Referencias
Atanasova, L., Jaklitsch, W. M., Komon-Zelazowska, M., Kubicek, C. P., & Druzhinina, I. S. (2010). Clonal species Trichoderma parareesei sp. nov. likely resembles the ancestor of the cellulase producer Hypocrea jecorina/T. reesei. Applied and Environmental Microbiology, 76(21), 7259–7267. http://doi.org/10.1128/AEM.01184-10
Bidochka, M. J., Kamp, A. M., Lavender, T. M., Dekoning, J., & De Croos, J. N. A. (2001). Habitat Association in Two Genetic Groups of the Insect-Pathogenic Fungus Metarhizium anisopliae: Uncovering Cryptic Species? Applied and Environmental Microbiology, 67(3), 1335–1342. http://doi.org/10.1128/AEM.67.3.1335-1342.2001
Castresana, J. (2000). Selection of conserved blocks from multiple alignments for their use in phylogenetic analysis. Molecular Biology and Evolution, 17(4), 540–552.
Chaverri, P., Branco-Rocha, F., Jaklitsch, W., Gazis, R., Degenkolb, T., & Samuels, G. J. (2015). Systematics of the Trichoderma harzianum species complex and the re-identification of commercial biocontrol strains. Mycologia, 107(3), 558–590. http://doi.org/10.3852/14-147
Chaverri, P., Castlebury, L. A., Samuels, G. J., & Geiser, D. M. (2003). Multilocus phylogenetic structure within the Trichoderma harzianum/Hypocrea lixii complex. Molecular Phylogenetics and Evolution, 27(2), 302–313. http://doi.org/10.1016/S1055-7903(02)00400-1
Consolo, V. F., Mónaco, C. I., Cordo, C. A., & Salerno, G. L. (2012). Characterization of novel Trichoderma spp. isolates as a search for effective biocontrollers of fungal diseases of economically important crops in Argentina. World Journal of Microbiology & Biotechnology, 28(4), 1389–1398. http://doi.org/10.1007/s11274-011-0938-5
Díaz, B. M., & Lecuona, R. E. (1995). Evaluación de cepas nativas del hongo entomopatógeno Beauveria bassiana Bals. (Vuill.) (Deuteromicotina) como base para la selección de bioinsecticidas contra el barrenador Diatraea saccharalis (F.). AgriScientia, 12(0), 33–38. http://doi.org/10.31047/1668.298x.v12.n0.2450
Druzhinina, I. S., Komoń-Zelazowska, M., Atanasova, L., Seidl, V., & Kubicek, C. P. (2010). Evolution and ecophysiology of the industrial producer Hypocrea jecorina (Anamorph Trichoderma reesei) and a new sympatric agamospecies related to it. PLoS ONE, 5(2), e9191–15. http://doi.org/10.1371/journal.pone.0009191
French, E. R., & Herbert, T. (1980). Métodos de investigación fitopatología. San José, Costa Rica: Editorial IICA.
Guigón-López, C., Guerrero-Prieto, V., Vargas-Albores, F., Carvajal-Millán, E., Ávila-Quezada, G. D., Bravo-Luna, L., Roucco, M., Lanzuise, S., Woo, S., Lorito, M. (2010). Identificación molecular de cepas nativas de Trichoderma spp. su tasa de crecimiento in vitro y antagonismo contra hongos fitopatogenos. Revista Mexicana De Fitopatología, 28(2), 87–96.
Howell, C. R. (2003). Mechanisms employed by Trichoderma species in the biological control of plant diseases: the history and evolution of current concepts. Plant Disease, 87(1), 4–10. http://doi.org/10.1094/PDIS.2003.87.1.4
Hoyos-Carvajal, L., Chaparro, P., Abramsky, M., Chet, I., & Orduz, S. (2008). Evaluation of Trichoderma spp. isolates against Rhizoctonia solani and Sclerotium rolfsii under in vitro and greenhouse conditions. Agronomía Colombiana, 26(3), 451–458.
Huson, D.H. (1998). SplitsTree: a program for analyzing and visualizing evolutionary data. Bioinformatics 14(1), 68–73.
Infante, D., Martínez, B., González, N., & Reyes, Y. (2009). Mecanismos de acción de Trichoderma frente a hongos fitopatógenos. Revista De Protección Vegetal, 24(1), 14–21.
Jaimes Suárez, Y. Y., Moreno Velandia, C. A., & Cotes Prado, A. M. (2009). Inducción de resistencia sistémica contra Fusarium oxysporum en tomate por Trichoderma koningiopsis Th003. Acta Biológica Colombiana, 14(3), 111–120.
Martínez, B., Infante, D., & Peteira, B. (2015). Taxonomía polifásica y variabilidad en el género Trichoderma. Revista De Protección Vegetal, 30, 11–22.
Mukerji, K. G., Manoharachary, C., & Singh, J. (Eds.). (2006). Microbial activity in the rhizosphere (Vol. 7). Berlin Heidelberg, Germany: Springer-Verlag. http://doi.org/10.1007/3-540-29420-1
Oksanen, J., Blanchet, F. G., Kindt, R., Legendre, P., McGlinn, D., Minchin, P. R., et al., (2017). vegan: Community Ecology Package. R package version 2.4-2. Recuperado de https://CRAN.R-project.org/package=vegan
Pal, K. K., & McSpadden, B. (2006). Biological control of plant pathogens. The Plant Health Instructor, 2, 1117–1142. http://doi.org/10.1094/PHI-A-2006-1117-02
Peakall, R., & Smouse, P. E. (2006). GENALEX 6: genetic analysis in Excel. Population genetic software for teaching and research. Molecular Ecology Notes, 6(1), 288–295. http://doi.org/10.1111/j.1471-8286.2005.01155.x
R Core Team. (2016). R: A language and environment for statistical computing. Vienna, Austria: Foundation for Statistical Computing. Retrieved from URL https://www.R-project.org/
Samuels, G. J., Ismaiel, A., de Souza, J., & Chaverri, P. (2012). Trichoderma stromaticum and its overseas relatives. Mycological Progress, 11(1), 215–254. http://doi.org/10.1007/s11557-011-0743-4
Sánchez, A. D., Barrera, V., Reybet, G. E., & Sosa, M. C. (2015). Biocontrol con Trichoderma spp. de Fusarium oxysporum causal del “mal de almácigos” en pre y post emergencia en cebolla. Revista De La Facultad De Agronomía, La Plata, 114(1), 61–70.
Stamatakis, A., Ludwig, T., & Meier, H. (2005). RAxML-III: a fast program for maximum likelihood-based inference of large phylogenetic trees. Bioinformatics, 21(4), 456–463. http://doi.org/10.1093/bioinformatics/bti191
Stayaert, J., Weld, R., Mendoza, A., Kryštofová, S., Šimkovič, M., Varečka, L., & Stewart, A. (2013). Asexual Development in Trichoderma. In P. K. Mukherjee, B. A. Horwitz, U. S. Singh, M. Mukherjee, & M. Schmoll (Eds.), Trichoderma (pp. 87–109). Oxfordshire, UK: CABI International.
Tapsoba, H., & Wilson, J. P. (1997). Effects of temperature and light on germination of urediniospores of the pearl millet rust pathogen, Puccinia substriata var. indica. Plant Disease, 81(9), 1049–1052. http://doi.org/10.1094/PDIS.1997.81.9.1049
Valencia-Abello, J. C., & Castro-Caicedo, B. L. (2004). Aspectos biológicos de aislamientos de Trichoderma sp. antagónicos a Rosellinia bunodes. Cenicafé, 55(1), 16–28.
Vargas, E. (2011). Guía para la identificación y manejo integrado de plagas en piña. Proagroin. Recuperado de https://docplayer.es/storage/25/5321315/1541796562/zQwkrWdAJ-XxDnEdAt7vFQ/5321315.pdf
Vásquez Jiménez, J. (2009). Evaluación de la eficacia in vitro de sustancias químicas y microorganismos antagónicos del género Trichoderma ssp., como herramienta para la toma de decisiones en el control de enfermedades “caso muerte descendente del cultivo de piña Ananas comosus (L) Merr.” San Carlos, Costa Rica.
Verma, P. C., Chakrabarty, D., Jena, S. N., & Mishra, D. K. (2009). The extent of genetic diversity among Vanilla species: Comparative results for RAPD and ISSR. Industrial Crops and Products, 29(2-3), 581–589. http://doi.org/10.1016/j.indcrop.2008.11.006
Watanabe, S., Kumakura, K., Kato, H., Iyozumi, H., Togawa, M., & Nagayama, K. (2005). Identification of Trichoderma SKT-1, a biological control agent against seedborne pathogens of rice. Journal of General Plant Pathology, 71(5), 351–356. http://doi.org/10.1007/s10327-005-0217-0
Wickham, H. (2016). ggplot2. AG Switzerland: Springer International Publishing.
7.Apéndices
Apéndice 1. Códigos y procedencia de muestras de Trichoderma aisladas de suelo en zonas boscosas en la zona norte de Costa Rica
|
Código |
Especie |
Zona de recolección |
|
8a |
T. reesei |
Río Cuarto |
|
9a |
T. koningiopsis |
Río Cuarto |
|
11fa |
T. koningiopsis |
Río Cuarto |
|
11fb |
T. asperellum |
Río Cuarto |
|
13b |
T. asperellum |
Pital de San Carlos |
|
1c |
T. koningiopsis |
Pital de San Carlos |
|
11e |
T. spirale |
Sarapiquí |
|
13c |
T. spirale |
Sarapiquí |
|
13a |
T. spirale |
Sarapiquí |
|
H1 |
T. asperellum |
Sarapiquí |
|
11c |
T. koningiopsis |
Muelle de San Carlos |
|
6a |
T. koningiopsis |
Muelle de San Carlos |
|
Ia |
T. koningiopsis |
Muelle de San Carlos |
1 Ingeniero agrónomo, Escuela de Ciencias Agrarias, Universidad Nacional (UNA), Costa Rica, josueucastro@gmail.com
2 Investigadora, Escuela de Ciencias Agrarias, Universidad Nacional (UNA), Costa Rica, steffany.orozco.cayasso@una.ac.cr
3 Especialista en biología molecular y biotecnología, Laboratorio de Análisis Genómico, Escuela de Ciencias Biológicas, Universidad Nacional (UNA), Costa Rica, rodolfo.umana.castro@una.ac.cr, ORCID 0000-0003-0041-2788
4 Especialista en genética hortícola, Escuela de Ciencias Agrarias, Universidad Nacional (UNA), Costa Rica, ramon.molina.bravo@una.ac.cr, ORCID 0000-0001-5564-4426



