
Rev. Ciencias Veterinarias, Vol. 40, N° 2, [1-8], E-ISSN: 2215-4507, julio-diciembre, 2022
DOI: https://doi.org/10.15359/rcv.40-2.1
URL: http://www.revistas.una.ac.cr/index.php/veterinaria/index
Evidencia de Circovirus tipo 3 (PCV3) en una granja suina de Costa Rica.
Evidence of porcine circovirus type 3 (PCV3) in a swine farm in Costa Rica.
Evidência de Circovirus tipo 3 (PCV3) em uma granja de suínos na Costa Rica.
Laura Chaverri1 , Fernando Chacón2, Evelyn Espinach Roel3, & Daniel Zeledón4
, Fernando Chacón2, Evelyn Espinach Roel3, & Daniel Zeledón4
1. Universidad Nacional, Escuela Medicina Veterinaria, Lagunilla, Heredia, Costa Rica,
laura.chaverri.esquivel@una.cr
2. Consultor, Cartago, Costa Rica, fchacon@porcinaamericana.com
3. Porcina Americana, Cartago, Costa Rica, eespinach@porcinaamericana.com
4. Laboratorio Diagnóstico molecular CGAT, Heredia, Costa Rica, danizele@yahoo.com
 Autora de correspondencia: laura.chaverri.esquivel@una.cr
Autora de correspondencia: laura.chaverri.esquivel@una.cr
Recibido: 8 de setiembre de 2020 Corregido: 11 de mayo de 2022 Aceptado: 13 de mayo de 2022
Resumen
Se reporta, por primera vez en Costa Rica, en una granja porcina ubicada en la provincia de Cartago, la presencia del patógeno Circovirus Porcino3 (PCV3). Se utilizó la técnica de PCR en Tiempo Real. De un total de 56 pooles de muestras de sangre, provenientes de animales con sintomatología respiratoria y desmedro, 32 resultaron positivos (57,14%) y 24 negativas (42.86%) para PCV3. Solo hubo un caso positivo para PCV2 y negativo para PCV3. Es importante dar seguimiento a la presencia de esta enfermedad en el país, con el fin de conocer su epidemiología e impacto en el hato porcino nacional.
Palabras clave: PCR tiempo real, Circovirus porcino 3 (PCV3), porcinos, Costa Rica.
Abstract
The presence of the pathogen Porcine Circovirus 3 (PCV3) is reported for the first time in Costa Rica in a swine farm located in the province of Cartago. The Real Time PCR technique was used. From a total of 56 pools of blood samples from animals with respiratory symptomatology and emaciation, 32 tested positive (57.14%) for PCV3, while 24 tested negative (42.86%). Only one case tested positive for PCV2 and negative for PCV3. The presence of this disease in the country must be monitored in order to know its epidemiology and its possible impact on the national swine herd.
Keywords: Real Time PCR, Porcine Circovirus 3 (PCV3), Porcine, Costa Rica.
Resumo
A presença do patógeno Circovírus Porcino 3 (PCV3) é relatada pela primeira vez na Costa Rica, em uma granja de suínos localizada na província de Cartago. Foi utilizada a técnica de PCR em Tempo Teal. De um total de 56 amostras de sangue de animais com sintomatologia respiratória e emagrecimento, 32 foram positivas (57,14%) para PCV3, enquanto 24 foram negativas (42,86%). Apenas um caso testou positivo para PCV2 e negativo para PCV3. A presença desta doença no país deve ser monitorada para conhecer sua epidemiologia e seu possível impacto no rebanho suíno nacional.
Palavras-chave: PCR em Tempo Real, Circovírus Porcino 3 (PCV3), suínos, Costa Rica.
1. Introducción
El circovirus porcino (PCV) es un virus desnudo, con genoma ADN monocatenario circular, perteneciente al género circovirus de la familia Circoviridae (Mankertz et al., 2004). Hasta ahora, se ha reportado tres especies de circovirus en cerdos: a) circovirus porcino 1 (PCV1), el cual no causa manifestaciones clínicas (Cheng, 2017) pero induce una respuesta inmune con anticuerpos en individuos infectados (Mankertz et al., 2004); b) circovirus porcino 2 (PCV2), el cual es más común en cerdos. Causa el síndrome emaciante multisistémico post destete, enfermedad respiratoria y entérica, dermatitis porcina, síndrome nefropático y falla reproductiva (Ouyang et al., 2019) y c) circovirus porcino 3 (PCV3), descrito recientemente, en apariencia, con amplia distribución (Kwon et al., 2018).
El PCV3 se reportó, por vez primera, en 2016 en Estados Unidos; luego en: Japón, Corea del Sur, China, Suecia, Rusia, Tailandia, Brasil, Dinamarca, Polonia, Italia y España (Franzo et al., 2018b; Ku et al., 2017; Kwon et al., 2017; Palinski et al., 2017; Shen et al., 2017; Stadejek et al., 2017, Tochetto et al., 2018). En Colombia, la presencia de PCV3 se informa, por primera vez, a partir de muestras de campo de cerdos con síntomas clínicos compatibles con PCV2 (Sun et al., 2019; Vargas-Bermúdez et al., 2019).
El PCV3 se divide en 3 grupos: PCV3a, PCV3b y PCV3c (Franzo et al., 2018b; Fux et al., 2018). Las cepas de PCV3 comparten una alta homología; por ejemplo, un 94.44 – 100% a nivel de nucleótidos y un 96.3-100% a nivel de aminoácidos (Geng et al., 2019).
En China, el PCV3a se subdivide en: PCV3a-1 y PCV3a-2; y una cepa intermedia PCV3a-IM. Por tanto, la diversidad genética de PCV3 tiende a incrementar a nivel mundial (Chen et al., 2019; Ouyang et al., 2019; Qi et al., 2019; Saraiva et al., 2019; Vargas-Bermúdez et al., 2019).
El PCV3 se ha detectado, mediante PCR, en: caninos, bovinos y ratones (Jiang et al., 2019). Cuatro cepas de PCV3, aisladas de ratones, fueron secuenciadas y comparadas con las secuencias del marco de lectura 2 (ORF2) y, del genoma completo de PCV3, aisladas de cerdos, observándose una homología de 97.9%-98.8% a nivel del ORF2 y de 96.9%-98.3% a nivel del genoma completo, lo cual sugiere transmisión entre especies (Jiang et al., 2019).
Se considera que los animales silvestres juegan un papel importante como reservorios potenciales del virus (Liu et al., 2019 citado en Ouyang et al., 2019). Que el PCV3 podría transmitirse a huéspedes no porcinos por rutas de transmisión entre especies (Sun et al., 2019).
Fux et al. (2018), en Alemania, reportan alta prevalencia del PCV3. Mediante análisis filogenético identificaron dos grupos de cepas del virus, las cuales se consideran genotipos. Según los autores, las posiciones específicas de los marcadores de nucleótidos y aminoácidos, en el ORF 1 y 2, deberían ser útiles para la clasificación, intraespecies y el genotipo de las cepas de PCV3. Adicionalmente, los mismos autores no encontraron una correlación entre las variantes de PCV3 y el origen geográfico, observando que la variabilidad hallada en Alemania es similar a la de otros países europeos, lo cual hace suponer que PCV3 no es un patógeno emergente.
El ADN del PCV3 se ha encontrado en casi todos los tejidos y fluidos, tanto en animales sanos como enfermos: cerebro, riñón, corazón, bazo, suero, efusión pleural, cavidad peritoneal, fluidos orales y nasales, heces, semen (Franzo et al., 2019; Ouyang et al., 2019) y calostro (Kedkovid et al., 2018).
La transmisión del PCV3 puede darse de manera horizontal o vertical. Además, el virus puede estar presente en cerdos aparentemente sanos o en animales enfermos (Kedkovid et al., 2018; Zheng et al., 2017) así como en aquellos de diferentes edades; lechones con tan solo un día de nacidos o cerdos con 24 semanas cumplidas (Kedkovid et al., 2018; Kwon et al., 2017; Palinski et al., 2017; Stadejek et al., 2017; Sukmak et al., 2019).
Las infecciones por PCV3 se han asociado con la presencia de las siguientes condiciones: inflamación cardíaca y multisistémica (Phan et al., 2016), dermatitis y síndrome de nefropatía e insuficiencia reproductiva (Palinski et al., 2017; Tochetto et al., 2017; Wang et al., 2017), abortos, momias, fetos nacidos muertos (Ku et al., 2017; Palinski et al., 2017), pirexia y neumonía (Franzo et al., 2018a; Franzo et al., 2019; Kedkovid et al., 2018; Palinski et al., 2017; Phan et al., 2016; Shen et al., 2017), diarrea (Zhai et al., 2017), enfermedad respiratoria (Phan et al., 2016) y neurológica (Chen et al., 2019; Phan et al., 2016). En términos de presentación clínica, los problemas digestivos se relacionan en un 10.4% con la enfermedad causada por el PCV3 y las alteraciones respiratorias en un 26.6% (Ouyang et al., 2019).
Dado el interés por conocer la presencia de patógenos en nuestro medio, se describe el hallazgo de PCV3 en una granja porcina en Costa Rica, mediante el uso de la reacción en cadena de la polimerasa en tiempo real (RT-PCR, por sus siglas en inglés).
2. Materiales y métodos
2.1. Recolección de muestras y preparación
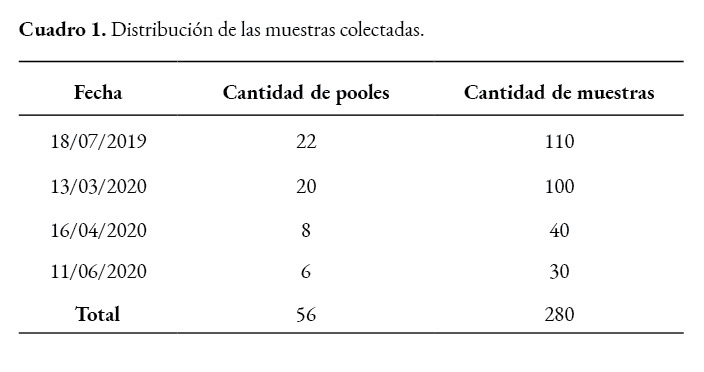
Se realiza cuatro muestreos en animales que presentan sintomatología respiratoria y desmedro, en una granja comercial de flujo continuo con 3000 vientres. Las muestras se colectan en distintos lotes de producción, los cuales tenían animales entre las cinco y veintitrés semanas de edad. La toma de muestras se lleva a cabo mediante venopunción de la vena yugular externa con una aguja calibre 21 x 1 ½; se recolectó cada una en tubos de ensayo sin anticoagulante y se transportaron refrigeradas al laboratorio para la elaboración de los pooles, cinco muestras por cada uno, para posterior análisis molecular.
2. 2. Extracción de ADN de muestras de suero porcino: análisis de la muestra, extracción y purificación de ADN mediante columnas.
La extracción y purificación del ADN total del suero de muestras de sangre porcina se realizó con el kit “Pure Link Genomic DNA Mini kit de Invitrogen” (N° de catálogo K1820-01) siguiendo las instrucciones del fabricante. El ADN extraído y purificado se almacenó a una temperatura de 2-8°C hasta su posterior análisis.
2.3. Análisis Mediante PCR Tiempo Real.
2. 3. 1. Cebadores y sonda
Los cebadores utilizados fueron fabricados por la casa comercial Invitrogen; la sonda por Applied Biosystems. Las secuencias de los cebadores y las sondas específicas, que se utilizan para PCV3, son las siguientes: primer forward: 5’CGGTGGGGTCATATGTGTTG-3’ (de 1443–1462 nucleótidos en GenBank no. NC-031753.1); primer reverse: 5’-CACAGCCGTTACTTCACC-3’ (de 1543–1560 nucleótidos en GenBank no. NC-031753.1); sonda: 5’ROX-CTTTGTCCTGGGTGAGCGCTGGTAG-BHQ2 3’ (de 1496–1520 nucleótidos en GenBank no. NC-031753.1) los cuales interrogan la región BMR83_gp1 en la posición 1336 a la 1980. El tamaño esperado del amplicón es de 118 bp. Thermo Fisher Scientific Inc. (2022).
Las secuencias de los cebadores y la sonda específicos que se utilizan para PCV2 son: primer forward: 5’ CCAGGAGGGCGTTSTGACT-3’ (de 1535–1553 nucleótidos en GenBank no. KX828228.1); primer reverse: 5’- CGYTACCGYTGGAGAAGGAA-3’ (de 1614–1633 nucleótidos en GenBank no. KX828228.1); sonda: 5 FAM-AATGGCATCTTCAACACCCGCCTCT-TAMRA 3 (de 1612–1592 nucleótidos en GenBank no. KX828228.1). Las cuales interrogan la región ORF2 en la posición 1030 a la 1734. El tamaño esperado del amplicón es de 99 bp. Thermo Fisher Scientific Inc. (2022).
2. 3. 2. Protocolo de PCR Tiempo Real para PCV2 y PCV3
Las pruebas se realizan en un termociclador de tiempo real 7500 de Thermofisher. Se tomaron 5 μL de ADN, extraído de la muestra, más ١٠ μL de TaqMan Universal Master Mix II (Marca Applied Biosystems, N° de catálogo 4440038) para un volumen total de reacción de 20μL. Los cebadores se reconstituyen a 100 micromolar, posteriormente, se utilizaron 400 nano molar para cada una de las reacciones (Kim et al., 2017). Se completó el volumen de la reacción con 2 μL de agua libre de nucleasas Ambio (N° de catálogo AM9938).
La amplificación se realizó en minitubos de PCR, con el siguiente perfil térmico: 3 minutos a 95°C seguido por 40 ciclos de 10 segundos a 95°C y 30 segundos de annealing y la extensión a 60°C se realiza la lectura de la fluorescencia. El valor se consideró negativo cuando el resultado fue superior a 37 ciclos; positivo cuando se obtuvo resultados inferiores a 37. Este valor se tomó como referencia de Kim et al. (2017).
Resultados y Discusión
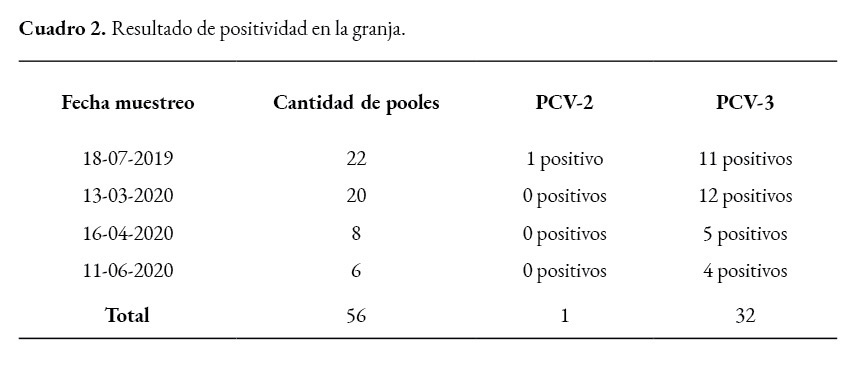
Se consideran positivos los valores inferiores a 37 ciclos en el RT-PCR, de un total de 56 pooles de sangre, 32 resultaron positivos (57,14%) y 24 (42,85%) negativos para PCV3, en cerdos con edades que oscilaban entre 5 y 23 semanas, con sintomatología respiratoria y desmedro. Únicamente un pool (1,78%) fue positivo a PCV2.
El resultado obtenido es superior a los reportados en Brasil y China en cerdos sintomáticos. Por ejemplo, en 9 estados de Brasil reportan un 47,8% (32/ 67) de PCV3, en los cuales la tasa de positividad en animales sanos fue de 29,8% (20/27); mientras que en los cerdos no sanos fue de 17.9% (12/67), lo cual sugiere que la mayoría de los cerdos puede vivir con la infección sin los síntomas clínicos (Saravia et al., 2019). Por otra parte; en China, se reporta una tasa de positividad a PCV3 de 31.18%, a nivel de animales, y de 48.78% (20/41) a nivel de granjas, oscilando el rango de 20% a 42.86% entre 2013 y 2017 (Xu PengLi et al., 2018).
Otro estudio, realizado en China, demostró que una alta tasa de detección de PCV-3, en el suero de cerdos asintomáticos, puede estar asociado con una baja tasa de replicación viral, que no conduce a inflamación detectable, lo cual podría ser un mecanismo de supervivencia del virus para evitar ser eliminado por el sistema inmune (Savic et al., 2019).
Asimismo, Savic et al. (2019) reportan que el PCV3 puede actuar como un agente secundario, que agrava la condición del animal, especialmente cuando sufre infección activa por PRRSV, enfermedad de Glasser, pasteurelosis y la enfermedad del complejo respiratorio porcino (PRDC por sus siglas en inglés).
A pesar de que en esta investigación no se evaluó posibles agentes concomitantes, presentes en la granja, es importante considerar, para futuros estudios, que las coinfecciones de PCV3, con otros patógenos, son muy comunes en los hatos porcinos. Por ejemplo, se han reportado muestras positivas de PCV3 con altas tasas de coinfección con PCV2, PRRSV, PEDV (virus de la diarrea epidémica porcina) y parvovirus porcino (Ouyang et al., 2019). Sin embargo, según Saravia et al. (2019), no hay correlación entre el PCV3, los signos clínicos y la interacción por PCV2.
Otros autores describen que la tasa de coinfección entre PCV3 y PCV2 es de 61.54%; en Corea reportan un 28.3% y en China 1.26 a 39.39% (Sukmak et al., 2019). Asimismo, se ha reportado una coinfección entre PCV3 y PRRSV de 61.54% (Chen et al., 2019; Zheng et al., 2018). En nuestro caso, únicamente se constató un pool positivo a PCV2 que resultó negativo para PCV3.
Conclusión
Se reporta, por primera vez, la presencia del patógeno denominado PCV3 en Costa Rica. Se recomienda dar seguimiento a la presencia de esta enfermedad en el país con el fin de conocer su epidemiología y posible impacto en el hato porcino nacional.
Los autores declaran que no hay conflicto de interés.
Referencias
Chen, GH., Mai, KJ., Zhou, L., Wu, RT., Tang, XY., Wu, JL., He, LL., Lan, T., Xie,QM., Sun, Y., & Ma, JY. (2017). Detection and genome sequencing of porcine circovirus 3 in neonatal pigs with congenital tremors in South China. Transbound Emerg Dis, 64(6):1650–1654. https://doi.org/10.1111/ tbed.12702
Chu, J., Guo, Y., Xi, Y., Song, H., Yuan, F., Wei, L., & Liu, J. (2019). Induction of porcine dermatitis and nephropathy syndrome in piglets by infection with porcine circovirus type 3. J Virol, 93. https://doi.org/10.1128/JVI.02045-18. pii: e02045-18
Jiang, S., Zhou, N., Li, Y., An, J., & Chang, T. (2019). Detection and sequencing of porcine circovirus 3 in commercially sourced laboratory mice. Vet Med Sci, 5, 176–181.
Franzo, G., Legnardi, M., Centelleghe, C., Tucciarone, C.M., Cecchinato, M., Cortey, M., Segalés, J. & Drigo, M. (2018a). Development and validation of direct PCR and quantitative PCR assays for the rapid, sensitive, and economical detection of porcine circovirus 3. J Vet Diagn Invest, 30, 538–544.
Franzo, G., Legnardi, M., Hjulsager, C.K., Klaumann, F., Larsen, L.E., Segales, J., & Drigo, M. (2018b). Full genome sequencing of porcine circovirus 3 field strains from Denmark, Italy and Spain demonstrates a high within-Europe genetic heterogeneity. Transbound Emerg Dis, 65, 602–606.
Franzo, G., Grassi, L., Tucciarone, C.M., Drigo, M., Martini, M., Pasotto, D., Mondin, A., & Menandro, M.L. (2019). A wild Circulation: high presence of Porcine circovirus 3 in different mammalian wild hosts and ticks. Transbound Emerg Dis, 66:1548–1557. https://doi.org/ 10.1111/tbed.13180
Fux, R., Söckler, C., Link, E. K., Renken, C., Krejci, R., Sutter, G., Ritzmann, M., & Eddicks, M. (2018). Full genome characterization of porcine circovirus type 3 isolates reveals the existence of two distinct groups of virus strains. Virology journal, 15(1), 25. https://doi.org/10.1186/s12985-018-0929-3
Geng, S., Luo, H., Liu, Y., Chen, C., Xu, W., Chen, Y., Li, X., & Fang, W. (2019). Prevalence of porcine circovirus type 3 in pigs in the southeastern Chinese province of Zhejiang. BMC Vet Res, 15: 244. https://doi.org/10.1186/s12917-019-1977-7
Kedkovid, R., Woonwong, Y., Arunorat, J., Sirisereewan, C., Sangpratum, N., Lumyai, M., Kesdangsakonwut, S., Teankum, K., Jittimanee, S., & Thanawongnuwech, R. (2018). Porcine circovirus type 3 (PCV3) infection in grower pigs from a Thai farm suffering from porcine respiratory disease complex (PRDC). Veterinary Microbiology, 215, 71-76.
Kim, HR., Park, YR., Lim, D., Park, MJ., Park, JY., Kim, SH., Lee, KK., Lyoo, YS., & Park, CK. (2017). Multiplex real-time polymerase chain reaction for the differential detection of porcine circovirus 2 and 3. J Virol Methods, 250, 11–16. https://doi.org/10.1016/j.jviromet.2017.09.021
Ku, X., Chen, F., Li, P., Wang, Y., Yu, X., Fan, S., & He, Q. (2017). Identification and genetic characterization of porcine circovirus type 3 in China. Transboundary and Emerging Diseases, 64, 703–708.
Kwon, T., Yoo, S.J., Park, C.K., & Lyoo, Y.S. (2017). Prevalence of novel porcine circovirus 3 in Korean pig populations. Vet Microbiol, 207, 178–180.
Li, X., Qiao, M., Sun, M., & Tian, K. (2018). A duplex realtime PCR assay for the simultaneous detection of porcine circovirus 2 and circovirus 3. Virol Sin, 33, 181–186.
Liu, X., Ouyang, T., Ouyang, H., Liu, X., Niu, G., Huo, W., Yin, W., Pang, D., & Ren, L., (2019). Human cells are permissive for the productive infection of porcine circovirus type 2 in vitro. Sci Rep, 9, 5638.
Mankertz, A., Caliskan, R., Hattermann, K., Hillenbrand, B., Kurzendoerfer, P., Mueller, B., Schmitt, C., Steinfeldt, T., & Finsterbusch, T. (2004). Molecular biology of Porcine circovirus: analyses of gene expression and viral replication. Veterinary Microbiology, 98 (2): 81-88. https://doi.org/10.1016/j.vetmic.2003.10.014
Opriessing, T., Xiao, C.T., Gerber, P.F., & Halbur, P.G. (2013). Emergence of a novel mutant PCV2b variant associated with clinical PCVAD in two vaccinated pig farms in the U.S. concurrently infected with PPV2. Vet Microbiol, 163, 177–83.
Opriessnig, T., Karuppannan, A.K, Castro, A.M.M.G., & Xiao, T.G. (2020). Porcine circoviruses: current status, knowledge gaps and challenges. Virus Research, Vol. 286. https://doi.org/10.1016/j.virusres.2020.198044
Ouyang, T., Niu, G., Liu, X., Zhang, X., Zhang, Y., & Ren, L. (2019). Recent progress on porcine circovirus type 3, Infection, Genetics and Evolution, https://doi.org/10.1016/j.meegid.2019.05.009
Palinski, R., Pineyro, P., Shang, P., Yuan. F., Guo, R., Fang, Y., & Hause, BM. (2017). A novel porcine circovirus distantly related to known circoviruses is associated with porcine dermatitis and nephropathy syndrome and reproductive failure. J Virol, 91, e01879-16. https:// doi.org/10.1128/JVI.01879-16 2
Phan, T.G., Giannitti, F., Rossow, S., Marthaler, D., Knutson, T.P., Li, L., Deng, X., Resende, T., Vannucci, F., & Delwart, E. (2016). Detection of a novel circovirus PCV3 in pigs with cardiac and multi-systemic infammation. Virol J,13(1):184. https://doi.org/10.1186/s1298 5-016-0642-z 3
Qi, S., Su, M., Guo, D., Li, C., Wei, S., Feng, L., & Sun, D. (2019). Molecular detection and phylogenetic analysis of porcine circovirus type 3 in 21 provinces of China during 2015-2017. Transbound Emerg Dis, 66, 1004–1015.
Saraiva, G.L., Pereira, P.M., Viviane, S.A., Murilo, L.M., Saraiva, A.N., Rangel, J.L., Costa, G., Portela, Z.I., De Almeida, M.R., & Silva, A. (2019). Retrospective Detection and Genetic Characterization of Porcine circovirus 3 (PCV3) Strains Identified between 2006 and 2007 in Brazil. Viruses, 11, 201. https://doi.org/10.3390/v11030201
Savic, B., Milicevic, V., Radanovic, O., Zdravkovic, N., Stevancevic, O., Kureljusic B., & Nesic, K. (2019). Identification and genetic characterization of porcine circovirus 3 on pig farms in Serbia, Archives of Virology, https://doi.org/10.1007/s00705-019-04455-y
Shen, H., Liu, X., Zhang, P., Wang, L., Liu, Y., Zhang, L., Liang, P., & Song, C. (2017). Genome characterization of a porcine circovirus type 3 in South China. Transbound. Emerg. Dis, 65(1): 264–266. https:// doi.org/10.1111/tbed.12639.
Stadejek, T., Wozniak, A., Miłek, D., & Biernacka K. (2017). First detection of porcine circovirus type 3 on commercial pig farms in Poland. Transboundary and Emerging Diseases, 64, 1350–1353. https://doi.org/ 10.1111/tbed.12672
Sukmak, M., Thanantong, N., Poolperm, P., Boonsoongnern, A., Ratanavanichrojn, N., Jirawattanapong, P., Woonwong, Y., Soda, N., Kaminsonsakul, T., Phuttapatimok, S., & Wajjwalku, W. (2019). The retrospective identification and molecular epidemiology of porcine circovirus type 3 (PCV3) in swine in Thailand from 2006 to 2017. Transbound. Emerg. Dis, 66, 611–616.
Sun, W., Wang, W., Xin, J., Cao, L., Zhuang, X., Zhang, C., Zhu, Y., Zhang, H., Qin, Y., Du, Q., Han, Z., Lu, H., Zheng, M., & Jin, N. (2019) An epidemiological investigation of porcine circovirus 3 infection in dogs in the Guangxi Province from 2015 to 2017, China, Virus Research. https://doi.org/10.1016/j.virusres.2019.197663
Thermo Fisher Scientific Inc. (2022). Invitrogen™ Minikit de ADN genómico PureLink™: N° de catálogo K1820-01. https://www.fishersci.es/shop/ products/invitrogen-purelink-genomic-dna-mini-kit-3/10053293
Tochetto, C., Lima, D.A., Varela, A.P.M., Loiko, M.R., Paim, W.P., Scheffer, C.M., Herpich, J.I., & Cerva, C. (2018). Full-genome sequence of porcine circovirus type 3 recovered from serum of sows with stillbirths in Brazil. Transbound Emerg Dis, 65, 5–9.
Vargas‐Bermudez, D., Campos, F., Bonil, L., Mogollon, D., & Jaime, J. (2019). First detection of porcine circovirus type 3 in Colombia and the complete genome sequence demonstrates the circulation of PCV3a1 and PCV3a2, Veterinary Medicine and Science, 5 (2) : 182-188. https://doi.org/10.1002/vms3.155
Xu, PengLi., Zhang, Yu., Zhao, Yu., Zheng, HuiHua., Han, HaoYing., Zhang, HongXin., Chen, HongYing., Yang, MingFan., & Zheng, LanLan. (2018). Detection and phylogenetic analysis of porcine circovirus type 3 in central China.Transboundary and Emerging Diseases, 65 (5) :1163-1169.
Zhai, S.L., Zhou, X., Zhang, H., Hause, B.M., Lin, T., Liu, R., Chen, Q.L., Wei, WK., Lv, D.H., Wen, X.H., Li, F., & Wang, D. (2017). Comparative epidemiology of porcine circovirus type 3 in pigs with different clinical presentations. Virol J, https://doi.org/10.1186/s1298 5-017-0892-4 4
Zheng S, J. Shi, X. Wu, Z. Peng, C. Xin, L. Zhang, Y. Liu, M. Gao, S. Xu, H. Han, J. Yu, W. Sun, X. Cong, J. Li, & Wang, J. (2017). Presence of Torque teno sus virus 1 and 2 in porcine circovirus 3‐positive pigs, Transboundary and Emerging Diseases, 65 (2): 327-330. https://doi.org/10.1111/tbed.12792
Zheng, S., Shi, J., Wu, X., Peng, Z., Xin, C., Zhang, L., Liu, Y., Gao, M., Xu, S., Han, H., Yu, J., Sun, W., Cong, X., Li, J., & Wang, J. (2018) Presence of Torqueteno sus virus 1 and 2 in porcine circovirus 3-positive pigs. Transbound Emerg Dis, 65(2):327-330. https://doi.org/10.1111/tbed.12792

Licencia Creative Commons Atribución-No-Comercial